SA-Med2D-20M是 SAM-Med2D 使用的数据集 ,该数据集由460万张医学图像和近2000万个对应的掩膜构成,涵盖了10种模态、31个主要器官和219个类别,是迄今为止最大的医学图像分割数据集。如今,我们在许可范围内开源了SA-Med2D-20M,欢迎大家规范使用数据,并向我们反馈意见(文末附讨论群加入方法)。

论文链接:
https://arxiv.org/abs/2311.11969
SAM-Med2D系列工作开源链接(点击“阅读原文”直达):
https://github.com/OpenGVLab/SAM-Med2D
数据下载链接:
https://openxlab.org.cn/datasets/GMAI/SA-Med2D-20M
PART.1
背景
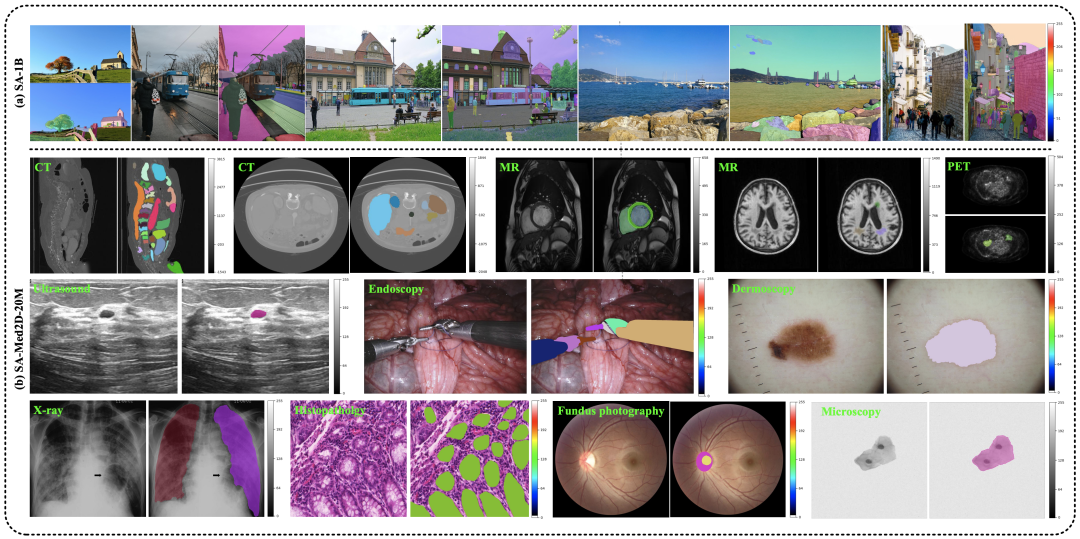
虽然没有单个大规模的医学图像数据集,但是海量的小而精的公开医学图像数据集为构建一个大规模医学图像数据集创造了条件。为了推进基础模型在医学图像分析中的发展,来自上海人工智能实验室通用视觉团队的研究者们通过收集和整理大量的公开和私人数据集后构建了一个超大规模的医学图像分割数据集: SA-Med2D-20M(图2),该数据集共有460万张医学图像和1970万个相应的掩膜,涵盖了10种模态、31个主要器官和219个类别,其分割目标覆盖了几乎全身,是一个大规模且具有数据多样性的医学图像分割数据集。
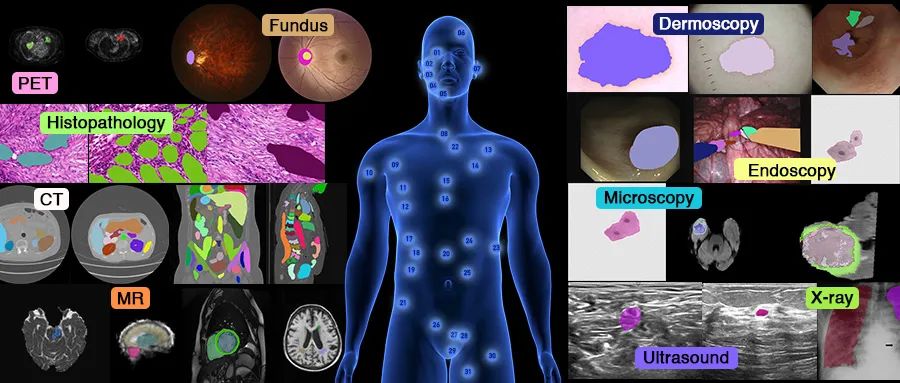
图2:SA-Med2D-20M由460万张医学图像和1970万个相应的掩膜组成
PART.2
SA-Med2D-20M数据集概览
2.1 子数据集信息统计
SA-Med2D-20M中的医学图像大部分来源于公开的医学分割数据,这些公开的数据集是从TCIA、OpenNeuro、NITRC、Grand Challenge、Synapse、CodaLab、GitHub等公开网络平台上获取的。按照这些数据集的病例(Case)数量和解剖结构情况可以对它们进行统计分析,其分布情况如图3所示。
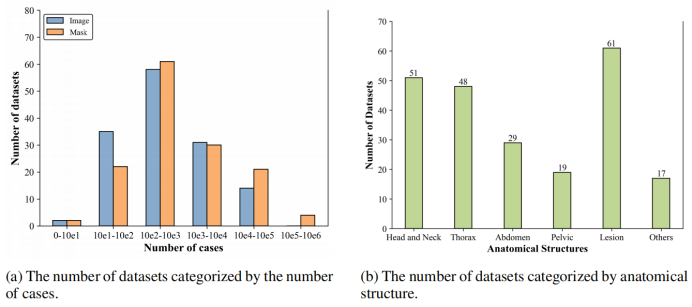
图3:所用到的子数据集的分布情况
2.2 模态信息统计
SA-Med2D-20M包括10种模态,详见表1,其分布如图4所示。CT和MR模态在图像和掩膜的数量上都占主导地位,这主要归因于它们在公共医学图像分割数据集中的广泛存在。此外,由于CT和MR扫描的是3D维度,研究者在三个轴上分别对其进行切分,这个过程会产生大量切片(Slice),这也会导致大量的图像和掩膜。继CT和MR之后,大多数模态的图像数量处于1,000到10,000之间,掩膜数量则处于1,000到20,000之间。值得注意的是,显微镜和组织病理学图像在该版本SA-Med2D-20M中各包含不到1,000张图像。研究团队计划在未来的数据集更新迭代中更加关注并扩增这些不太常见的模态数据。
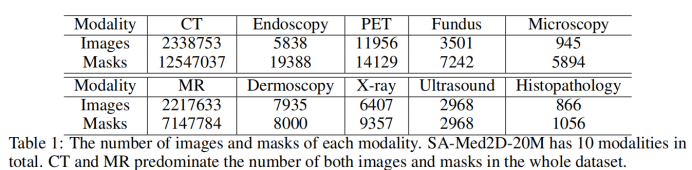 表1:不同模态的图像和掩膜数量
表1:不同模态的图像和掩膜数量
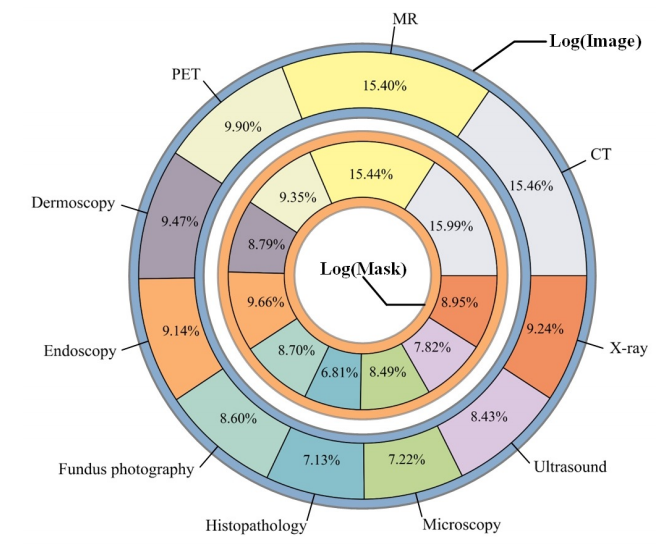 图4:SA-Med2D-20M的模态分布情况
图4:SA-Med2D-20M的模态分布情况
2.3 解剖结构信息统计
根据解剖结构和病变的存在,SA-Med2D-20M数据集被分为不同的类别,包括头颈部(Head and Neck)、胸部(Thorax)、腹部(Abdomen)、骨盆(Pelvic)、病变(Lesion)和其他(Others),如图5所示。各类别所包含的图像和掩膜数量如图6所示,其中,头颈部(Head and Neck)的图像和掩膜数量最多,因为该解剖结构存在大量多模态的脑部数据集,例如BraTS和ISLES系列。相比之下,其他(Others)类别包含的图像和掩膜最少。
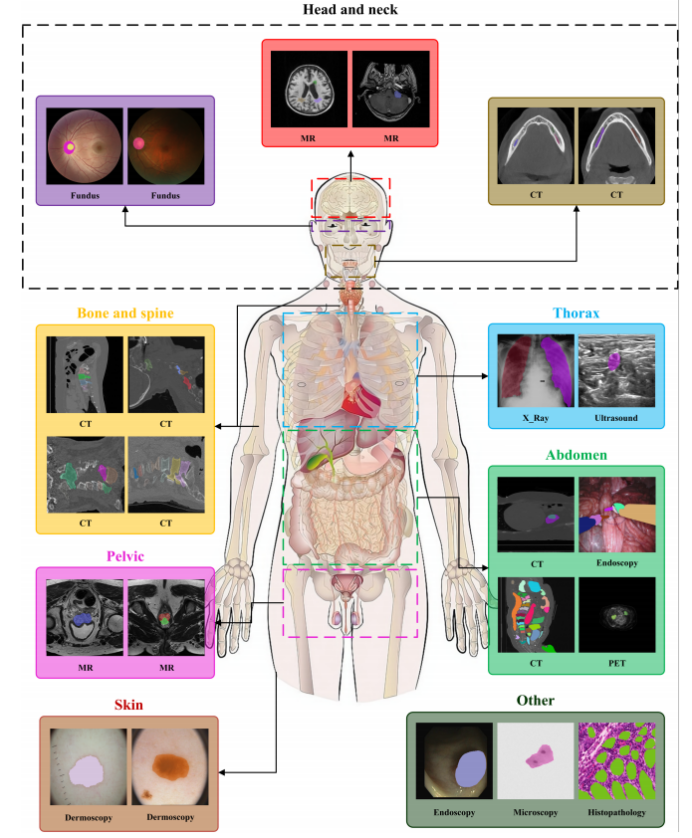 图5:SA-Med2D-20M所涵盖的解剖结构及各结构对应的示例图片
图5:SA-Med2D-20M所涵盖的解剖结构及各结构对应的示例图片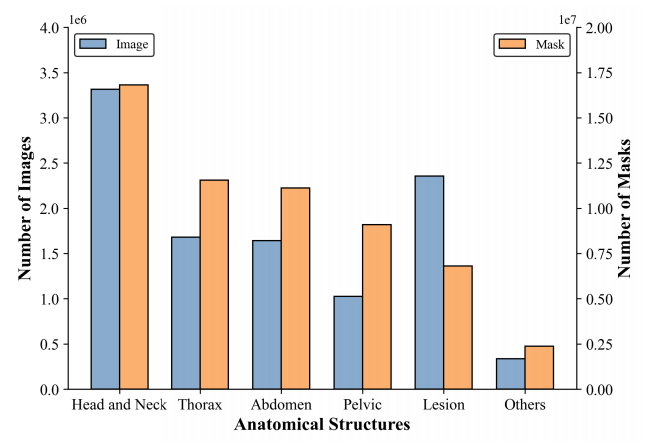 图6:各解剖结构的图像和掩膜数量
图6:各解剖结构的图像和掩膜数量2.4 类别信息统计
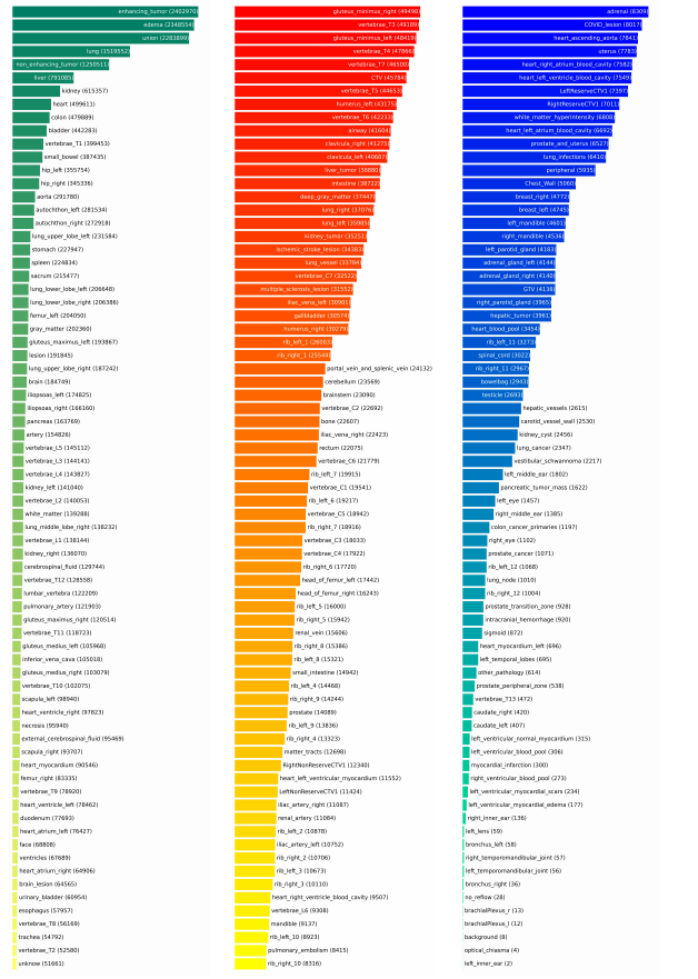
SA-Med2D-20M含219个类别标签,其详细分布如图7所示。总体来说SA-Med2D-20M的类别是长尾分布的。具体而言,47个类别的掩膜在100,000至1,000,000之间,51个类别的掩膜在1,000至10,000之间。最常见的范围是10,000至100,000个,共有88个类别。此外,还有28个类别的掩膜的数量少于1,000个。从具体的类别来看,增强性肿瘤(enhancing tumor)和水肿(edema)这两类的掩膜数量最多。联合(union)类别专门用于解决多个类别之间像素重叠的问题,这些类别的掩膜覆盖两个以上的类别。此外,标签未知(unknown)是指原始数据集未提供特定标签信息。
PART.3
SA-Med2D-20M数据集的构建流程
3.1 数据收集
构建SA-Med2D-20M的第一步是收集用于医学图像分割的数据集。研究团队首先从TCIA、OpenNeuro、NITRC、Grand Challenge、Synapse、CodaLab、GitHub等公开网络平台上收集大量的医学图像数据集。在收集海量数据后,需要对各个类型的数据集进行归档和统一的数据格式处理。具体包括了图像归一化、掩膜的拆分和合并等处理,最终通过统一的命名方式将处理好的图像和掩膜存为png格式的图片。
3.2 图像归一化
由于医学影像数据集的像素/体素的强度(intensity)差异较大,这会给模型的训练带来不利影响。因此,首先需要将不同模态的医学图像进行归一化。采用最大最小归一化的方式:
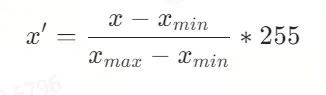
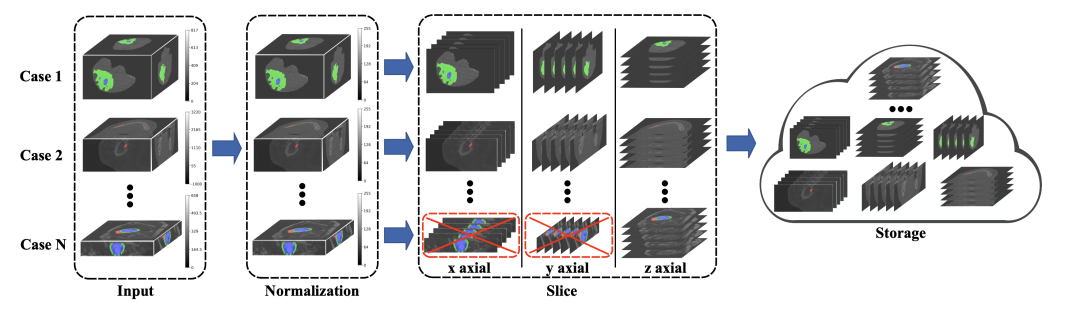 图8:图像归一化到存储的过程
图8:图像归一化到存储的过程3.3 掩膜的处理
研究者对掩膜的处理主要分为三步,图9显示了处理掩膜的整个流程:(1)将原始的语义掩膜拆分为二进制掩膜,这种方式与SAM类似。(2)进一步将二进制前景进行分离和合并的处理。分离是为了将具备多个联通域的类别拆分为多个单联通域目标,例如肺可以拆分为左肺和右肺;合并是在一个大的掩膜中合并其包含的其他掩膜,例如在一个肾中包含了病灶区域,通过第一步二进制拆分之后,肾区域将会出现一块中空区域,合并操作就是将这些中空区域进行补全。(3)去除不符合标准的掩膜。该步骤将去掉前景占比非常小的掩膜(0.153%)。
 图9:掩膜的处理流程
图9:掩膜的处理流程
PART.4
SA-Med2D-20M数据集的应用与展望

欢迎所有的合作,包括医院、研究院、高校、公司等。请联系
hejunjun@pjlab.org.cn
推荐阅读
本篇文章来源于微信公众号: 医工学人